A doença de Parkinson é marcada pela perda lenta e contínua de neurônios que produzem dopamina, um neurotransmissor que atua como um “lubrificante” químico, auxiliando nosso cérebro a coordenar movimentos. Uma das principais vilãs desse processo é a proteína α-sinucleína, que costuma ficar solta e inofensiva dentro das células, mas em certas circunstâncias se amontoa formando fibrilas tóxicas. Impedir esse ajuntamento sempre foi um desafio, porque identificar o contorno tridimensional da proteína que indica onde um medicamento pode se encaixar exigia meses de experimentos caros em laboratórios de cristalografia.
Até pouco tempo, determinar a forma de uma proteína era uma tarefa extremamente demorada. Primeiro, os cientistas precisavam produzir litros da proteína em bactérias ou células de mamífero e depois purificá-la até ficar quase 100% livre de impurezas. Em seguida, era necessário induzir a cristalização da proteína e obter pequenos cristais ordenados, tarefa delicada que podia exigir semanas de tentativas, pois cada proteína requer condições específicas de temperatura, sal e pH para se organizar. Quando o cristal finalmente crescia, ele era colocado diante de um feixe de raios X potente; os raios se espalhavam e geravam um padrão de pontinhos de luz num detector. Com base nesses pontinhos, computadores calculavam um mapa de densidade eletrônica que os pesquisadores precisavam interpretar átomo por átomo, ajustando um modelo 3D até ele “encaixar” no mapa. Todo esse percurso podia levar meses ou até anos e custava caro, exigindo equipamentos de grande porte.
Com o avanço das inteligências artificiais nos últimos anos, esse processo passou por uma grande mudança. Algoritmos como o AlphaFold 2 conseguem prever, em questão de minutos, a estrutura 3D da α-sinucleína apenas lendo a sequência de seus aminoácidos. É como ter um modelo digital da proteína pronto para inspeção, sem precisar cultivar cristais ou usar grandes máquinas de raios X. Com esse molde em mãos, programas de IA treinados em química compararam milhões de moléculas virtuais e calcularam quais delas se encaixariam melhor nos “bolsões” identificados na superfície da proteína. Assim, a triagem que antes exigia testar milhares de substâncias em laboratório passou a selecionar poucas dezenas de opções que pudessem cumprir o objetivo.

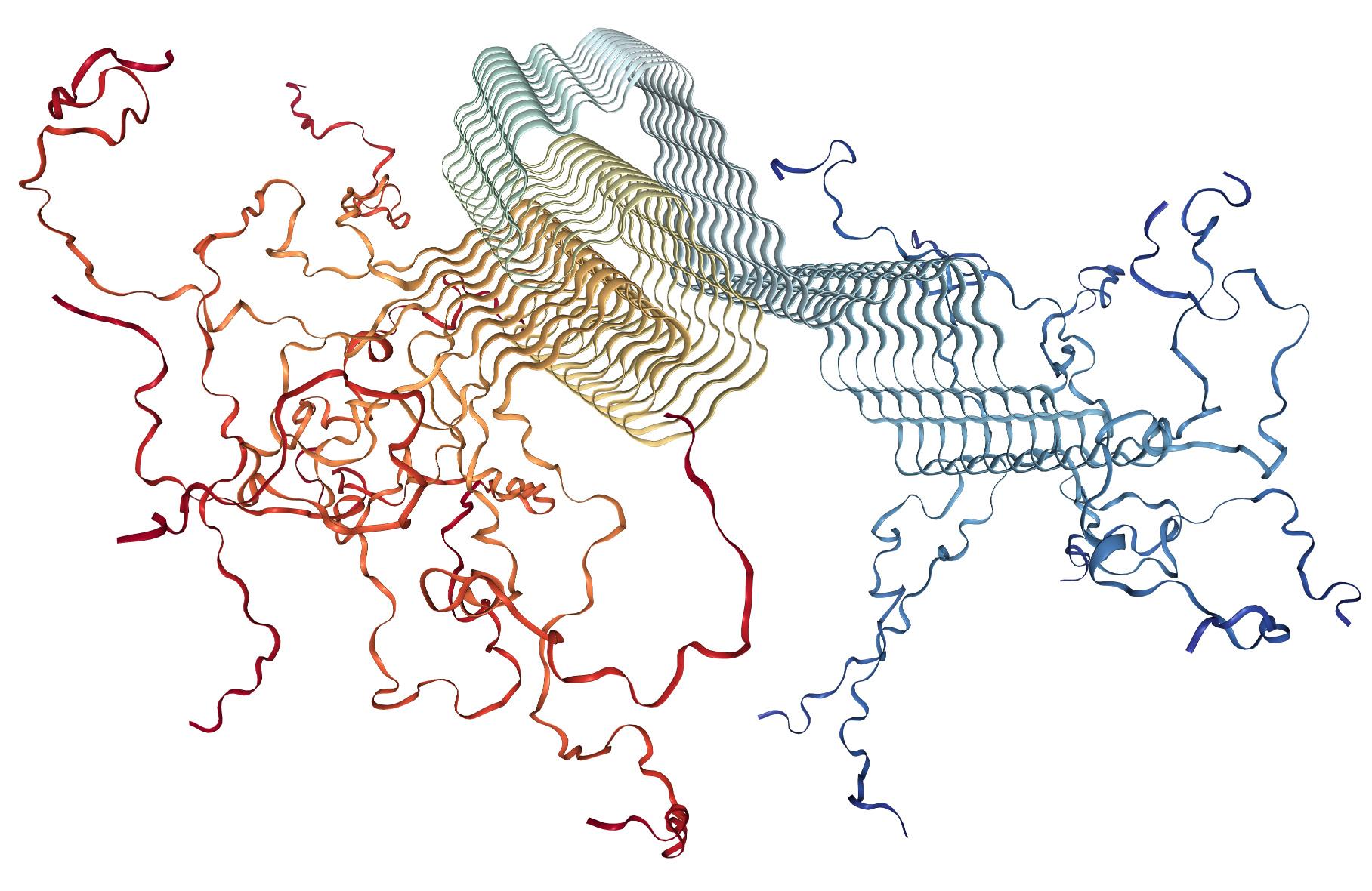
Estrutura tridimensional da α-sinucleína prevista pelo AlphaFold 2.
Fonte: Sino Biological, Alpha-Synuclein Protein Overview
Em 2024, um grupo da Universidade de Cambridge mostrou como essa abordagem ajuda diretamente na procura de remédios contra o Parkinson. Eles pegaram o modelo 3D da α-sinucleína (previsto pelo AlphaFold 2) e, como se fosse um mapa, marcaram os “buraquinhos” onde uma molécula candidata poderia se encaixar e impedir que a proteína se juntasse em placas. Depois, deixaram o computador “folhear” uma biblioteca digital com cinco milhões de moléculas, e em poucas horas, a inteligência artificial apontou quais pareciam caber melhor nesses buraquinhos. As dez melhores foram então sintetizadas em laboratório e testadas numa solução que contém α-sinucleína. Cinco delas funcionaram muito bem, reduzindo em cerca de 70% a formação das fibrilas tóxicas, mesmo quando usadas em quantidades minúsculas. Todo esse processo levou apenas algumas semanas e custou uma pequena fração do método tradicional, que exigiria testar substância por substância à mão. Esse exemplo mostra, de forma bem concreta, como conhecer a “forma” da proteína graças à IA encurta e barateia o caminho até possíveis medicamentos, como ilustrado na imagem a seguir.

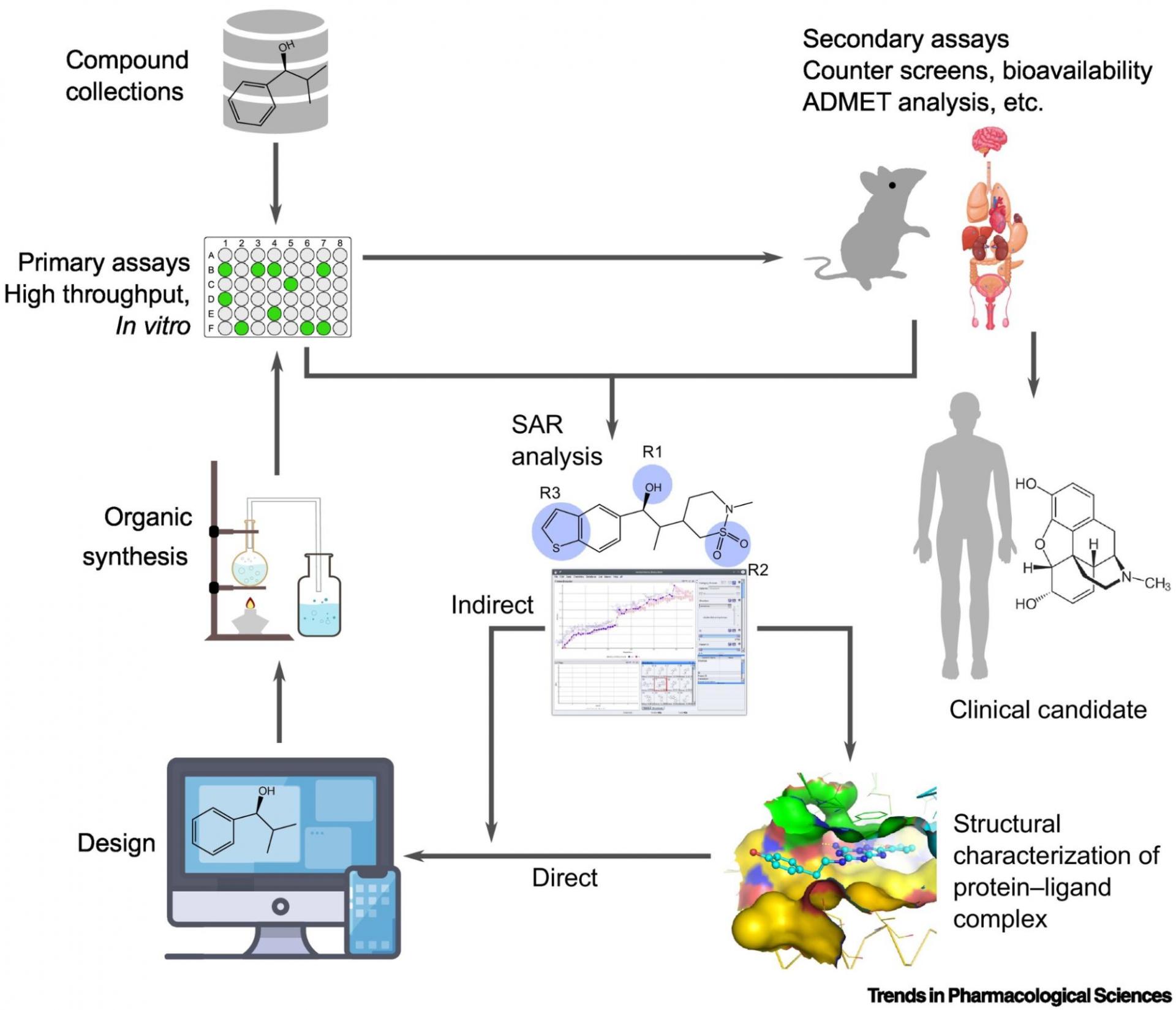
Pipeline de descoberta de fármacos guiado por inteligência artificial, do modelo computacional ao ensaio clínico.
Fonte: Zhang et al. (2020)
Entretanto, é necessário reforçar que a aceleração proporcionada pela inteligência artificial não dispensa o rigor científico. Cada resultado obtido no computador precisa ser confirmado por técnicas experimentais, que vão desde microscopia de alta resolução até ensaios celulares e testes em modelos animais, para garantir que o efeito previsto realmente se manifeste no organismo.
Ademais, há a preocupação com a questão das “caixas-pretas”, em que muitos algoritmos apresentam a solução sem revelar com clareza o raciocínio interno, dificultando compreender por que um candidato foi priorizado e outro descartado. Se aplicado o rigor ético necessário à sua utilização, a perspectiva geral é positiva, pois ao transformar o antigo método de tentativa e erro em uma busca guiada por modelos precisos, a IA acelera resultados, economiza recursos e aproxima a ciência de terapias verdadeiramente eficazes para a grande parcela da população que convive com o Parkinson.
Autor: Renan Bordignon Poy
Referências:
TUNYASUVUNAKOOL, K. et al. Highly accurate protein structure prediction for the human proteome. Nature, v. 596, p. 590-596, 22 jul. 2021. Disponível em: https://www.nature.com/articles/s41586-021-03828-1
BROTZAKIS, Z. F. et al. AlphaFold prediction of structural ensembles of disordered proteins. Nature Communications, v. 16, p. 1632, 14 fev. 2025. Disponível em: https://www.nature.com/articles/s41467-025-56572-9
UNIVERSITY OF CAMBRIDGE. AI speeds up drug design for Parkinson’s by ten-fold. ScienceDaily, 17 abr. 2024. Disponível em: https://www.sciencedaily.com/releases/2024/04/240417131018.htm.
KRISHNA, R. et al. Generalized biomolecular modeling and design with RoseTTAFold All-Atom. Science, v. 384, n. 6693, eadl2528, 19 abr. 2024. Disponível em: https://www.science.org/doi/10.1126/science.adl2528
BROWNE, G. DeepMind’s AI has finally shown how useful it can be. Wired, 22 jul. 2021. Disponível em: https://www.wired.com/story/deepmind-protein-folding-database/.
HORNE, R. I. et al. Discovery of potent inhibitors of α-synuclein aggregation using structure-based iterative learning. Nature Chemical Biology, v. 20, p. 634-645, 17 abr. 2024. Disponível em: https://doi.org/10.1038/s41589-024-01580-x.
CALLAWAY, E. AI protein-prediction tool AlphaFold 3 is now more open. Nature, v. 635, p. 531-532, 11 nov. 2024. Disponível em: https://www.nature.com/articles/d41586-024-03708-4
